
***tanggle***: Visualización de redes filogenéticas con *ggplot2*
Klaus Schliep
Graz University of TechnologyMarta Vidal-García
University of CalgaryLeann Biancani
University of Rhode IslandFrancisco Henao-Diaz
University of British ColumbiaEren Ada
University of Rhode IslandClaudia Solís-Lemus
University of Wisconsin-Madisonklaus.schliep@gmail.com Source:
vignettes/tanggle_vignette_espanol.Rmd
tanggle_vignette_espanol.RmdIntroducción
Esta es la viñeta en español para el paquete de R tanggle, en ella proveemos una vista general de sus funciones y ejemplos de uso. Tanggle extiende el paquete de R ggtree (Yu et al. 2017), lo cual permite la visualización de múltiples tipos de redes filogenéticas usando la sintaxis de ggplot2 (Wickham 2016). Especificamente, tanggle contiene funciones que permiten al usuario visualizar: (1) redes divididas o implícitas (no-enraizadas, no-direccionadas) y (2) redes explícitas (enraizadas, direccionadas) con reticulaciones. Estas funciones ofrecen alternativas a las funciones gráficas disponibles en ape (Paradis and Schliep 2018) y phangorn (Schliep 2011).
Lista de funciones
| name | Brief description |
|---|---|
geom_splitnet |
Adds a splitnet layer to a ggplot, to combine visualising data and the network |
ggevonet |
Grafica una red explícita de un objeto phylo |
ggsplitnet |
Grafica una red implícita de un objeto phylo |
minimize_overlap |
Reduce el número de líneas de reticulación entrecruzadas en la gráfica |
node_depth_evonet |
Devuelve las profundidades o alturas de los nodos y puntas en la red filogenética |
Para empezar
Instalar el paquete desde Bioconductor directamente:
Install the package from Bioconductor directly:
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install("tanggle")O instalar la versión de desarrollo del paquete desde: Github.
if (!requireNamespace("remotes", quietly=TRUE))
install.packages("remotes")
remotes::install_github("KlausVigo/tanggle")Si necesita installer ggtree desde github:
remotes::install_github("YuLab-SMU/ggtree")Y cargar todas las librerías:
Redes dividida o implicitas
Las redes divididas son objetos de visualización de datos que
permiten la definición de 2 (o más) opciones de división no compatibles.
Las redes divididas son usadas frecuentemente para graficar redes
consenso (Holland et al. 2004) o redes
vecinas (Bryant and Moulton 2004). Esto
puede llevarse a cabo utilizando las funciones consensusNet
o neighbor-net en phangorn (Schliep 2011), o importando archivos Nexus
provenientes de SplitsTree (Huson and Bryant
2006).
Tipos de datos
tanggle acepta tres formatos de entrada para redes divididas. Las siguientes opciones de entrada generan un objeto network para graficar.
Archivo Nexus creado con SplitsTree (Huson and Bryant 2006) e importado con la función
read.nexus.networken phangorn (Schliep 2011).Carga de red dividida en formato Nexus:
fdir <- system.file("extdata/trees", package = "phangorn")
Nnet <- phangorn::read.nexus.networx(file.path(fdir,"woodmouse.nxs"))- Una colección de árboles de genes (e.g., de RAxML (Stamatakis 2014)) en alguno de los siguientes
formatos: Importar archivo Nexus con la función
read.nexusArchivo de texto en formato Newick (un árbol de genes por línea) importado con la funciónread.tree
Estimación de una red dividida consenso mediante la función
consensusNet en phangorn (Schliep 2011).
- Secuencias en Nexus, Fasta o formato Phylip importandas con la
función
read.phyDaten phangorn (Schliep 2011) o la funciónread.dnaen ape (Paradis and Schliep 2018). Luego se calculan las matrices de distancia para los modelos de evolución específicos utilizando la funcióndist.mlen phangorn (Schliep 2011) odist.dnaen ape (Paradis and Schliep 2018). Con base en las matrices de distancia, se reconstruye una red dividida utilizando la funciónneighborNeten phangorn (Schliep 2011). Opcional: las longitudes de las ramas pueden ser estimadas utilizando la funciónsplitsNetworksen phangorn (Schliep 2011).
Para graficar una Red Dividida
Podemos graficar una red con las siguientes opciones por defecto:
p <- ggsplitnet(Nnet) + geom_tiplab2()
#> Warning: `aes_string()` was deprecated in ggplot2 3.0.0.
#> ℹ Please use tidy evaluation idioms with `aes()`.
#> ℹ See also `vignette("ggplot2-in-packages")` for more information.
#> ℹ The deprecated feature was likely used in the ggtree package.
#> Please report the issue at <https://github.com/YuLab-SMU/ggtree/issues>.
#> This warning is displayed once every 8 hours.
#> Call `lifecycle::last_lifecycle_warnings()` to see where this warning was
#> generated.
#> Warning: `aes_()` was deprecated in ggplot2 3.0.0.
#> ℹ Please use tidy evaluation idioms with `aes()`
#> ℹ The deprecated feature was likely used in the ggtree package.
#> Please report the issue at <https://github.com/YuLab-SMU/ggtree/issues>.
#> This warning is displayed once every 8 hours.
#> Call `lifecycle::last_lifecycle_warnings()` to see where this warning was
#> generated.
p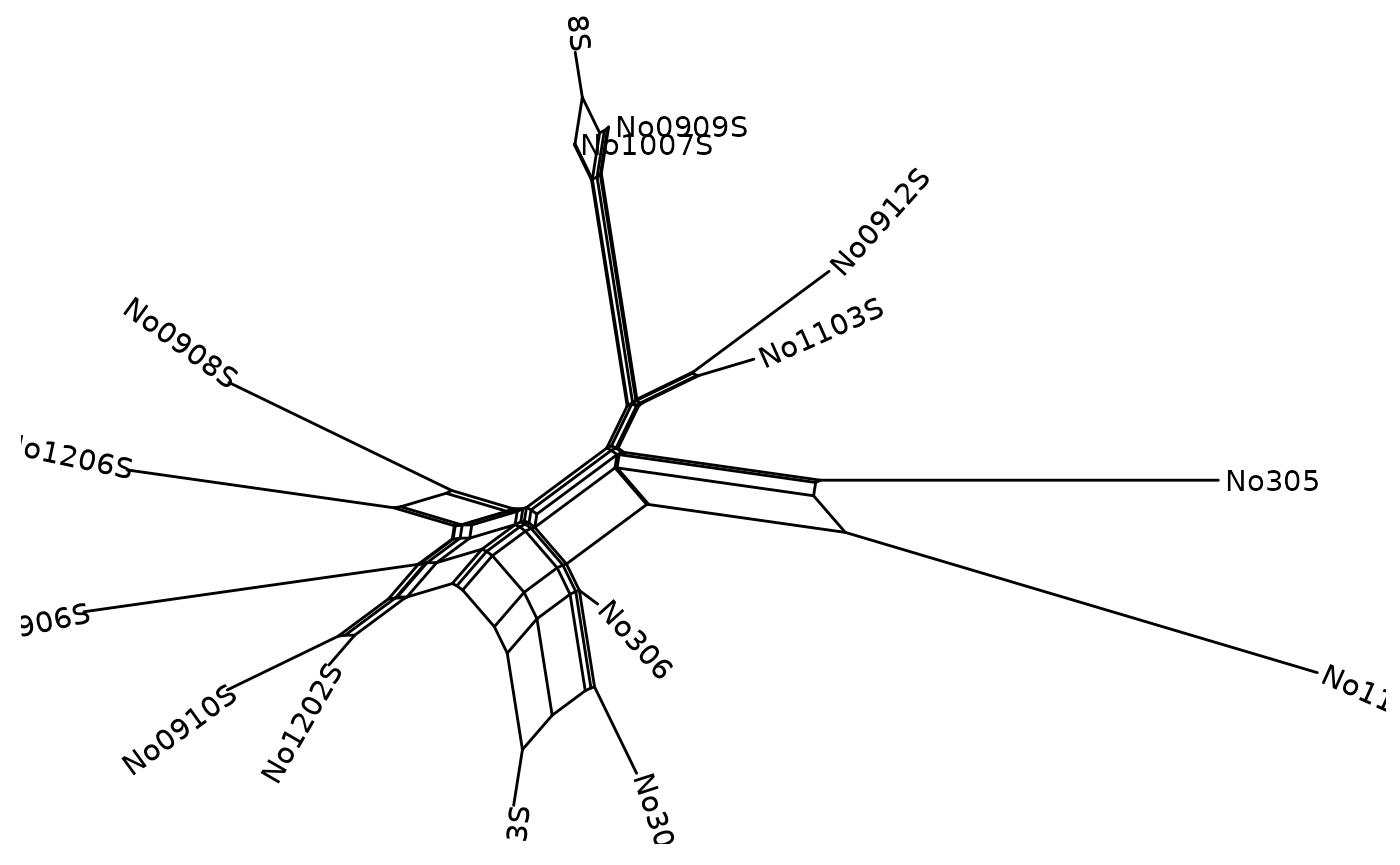
Luego podemos establecer los límites para los ejes x & y permitiendo la lectura de los nombres de los ejes.
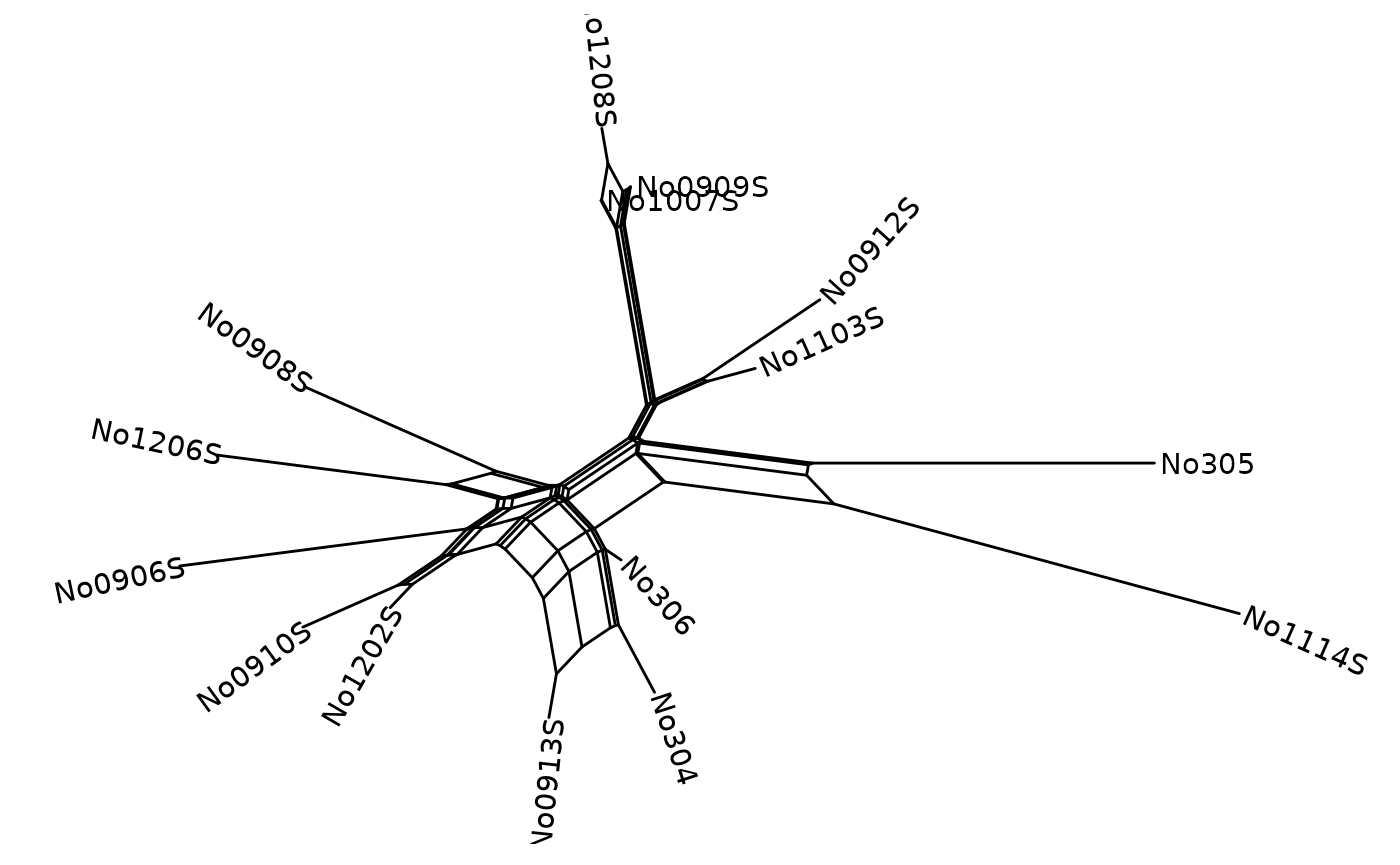
Es posible renombrar las puntas. Aquí cambiamos los nombres por un consecutivo de 1 a 15:
Nnet$translate$label <- seq_along(Nnet$tip.label)Podemos incluir los nombres de las puntas con
geom_tiplab2, y con esto personalizar algunas de sus
opciones. Por ejemplo, las puntas de color azul, en negrilla e itálicas;
también los nodos internos en verde:
ggsplitnet(Nnet) + geom_tiplab2(col = "blue", font = 4, hjust = -0.15) +
geom_nodepoint(col = "green", size = 0.25)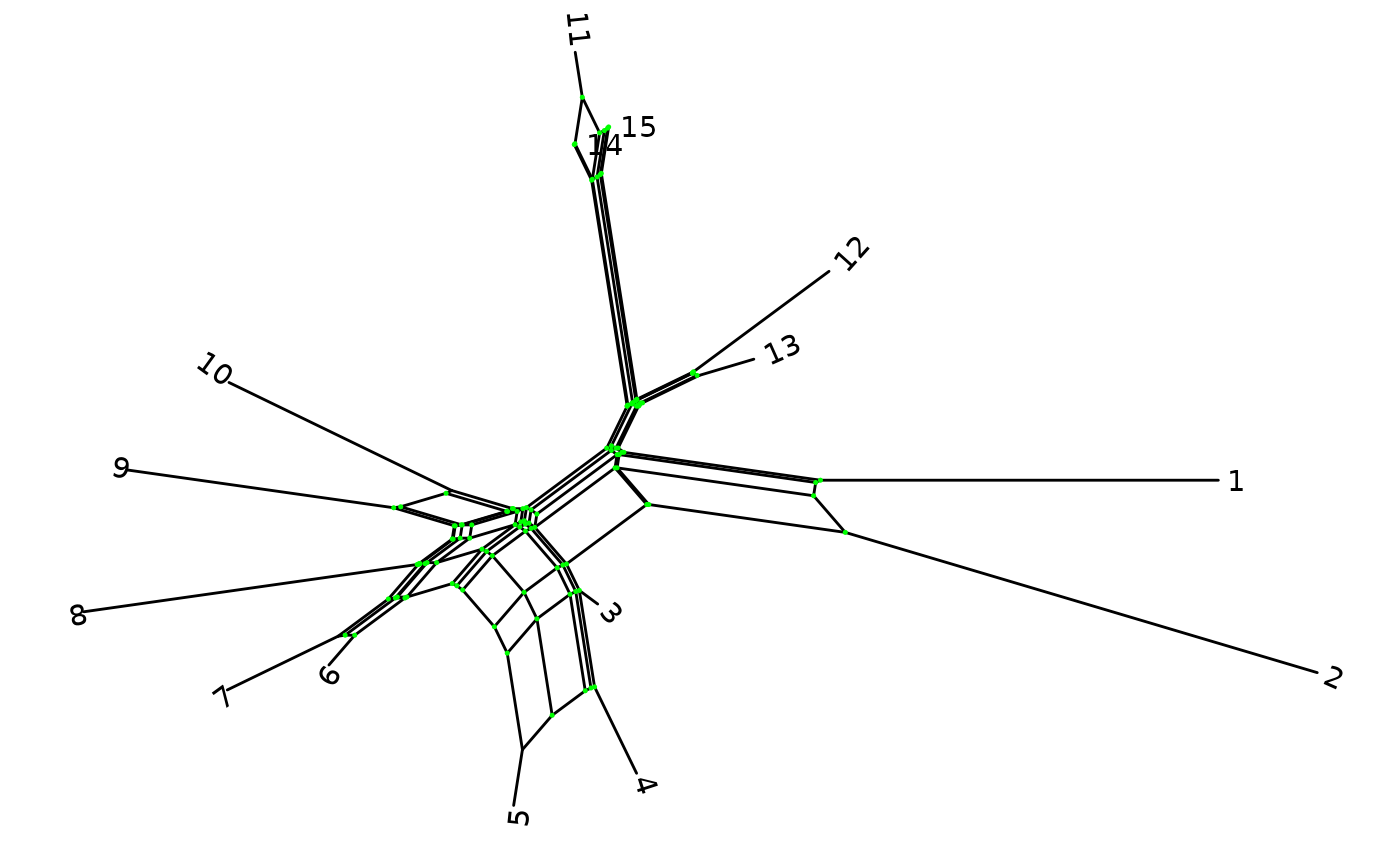
Los nodos pueden ser anotados con geom_point.
ggsplitnet(Nnet) + geom_point(aes(shape = isTip, color = isTip), size = 2)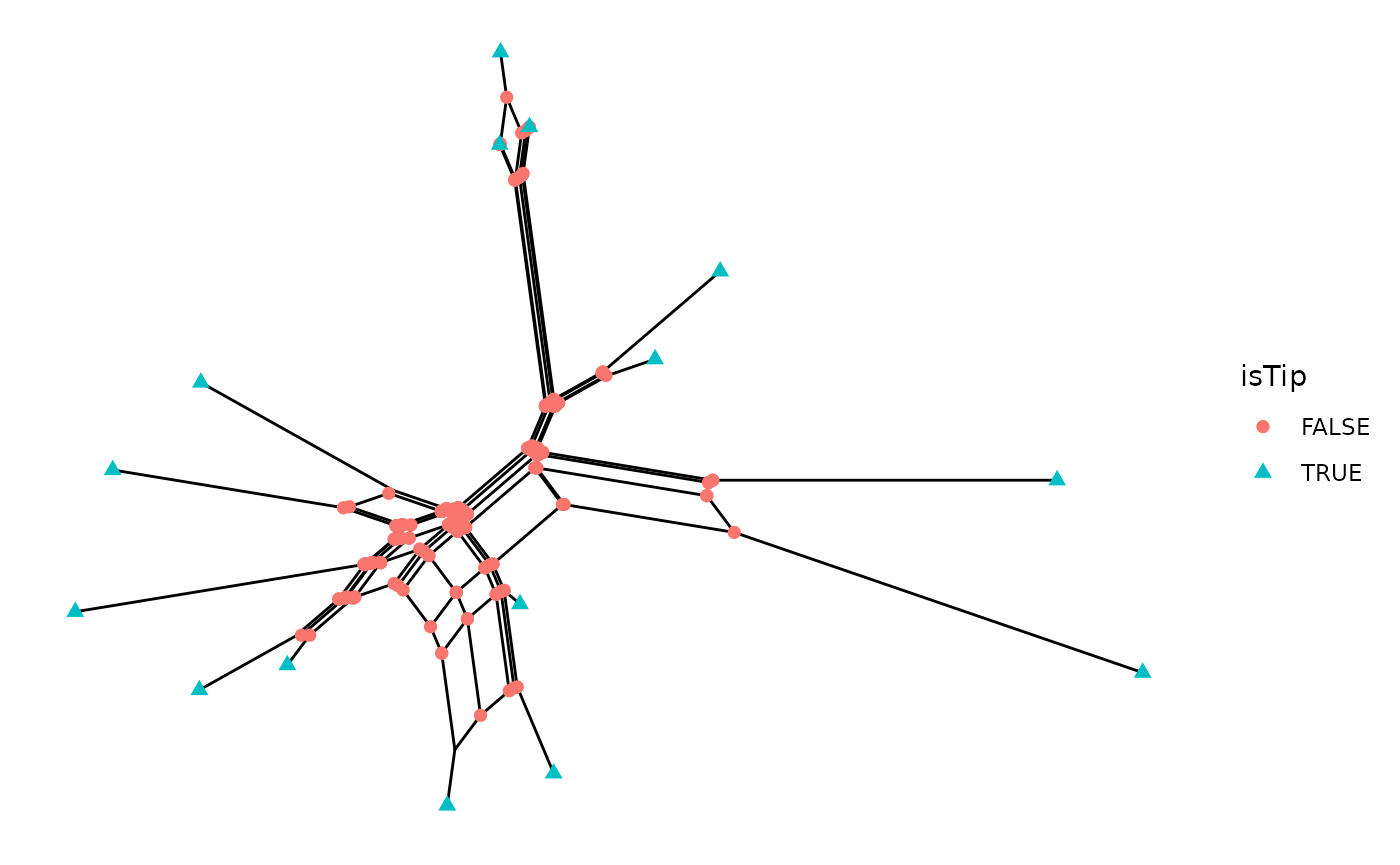
Para graficar una Red Explícita
La función ggevonet dibuja redes explícitas (árboles
filogenéticos reticulados). Una adición reciente en ape (Paradis and Schliep 2018) permite importar
árboles en un formato Newick extendido (Cardona,
Rosselló, and Valiente 2008).
Importar una red explícita (ejemplo de Fig. 2 en Cardona et al. 2008):
z <- read.evonet(text = "((1,((2,(3,(4)Y#H1)g)e,(((Y#H1,5)h,6)f)X#H2)c)a,
((X#H2,7)d,8)b)r;")Para graficar una red explícita:
ggevonet(z, layout = "rectangular") + geom_tiplab() + geom_nodelab()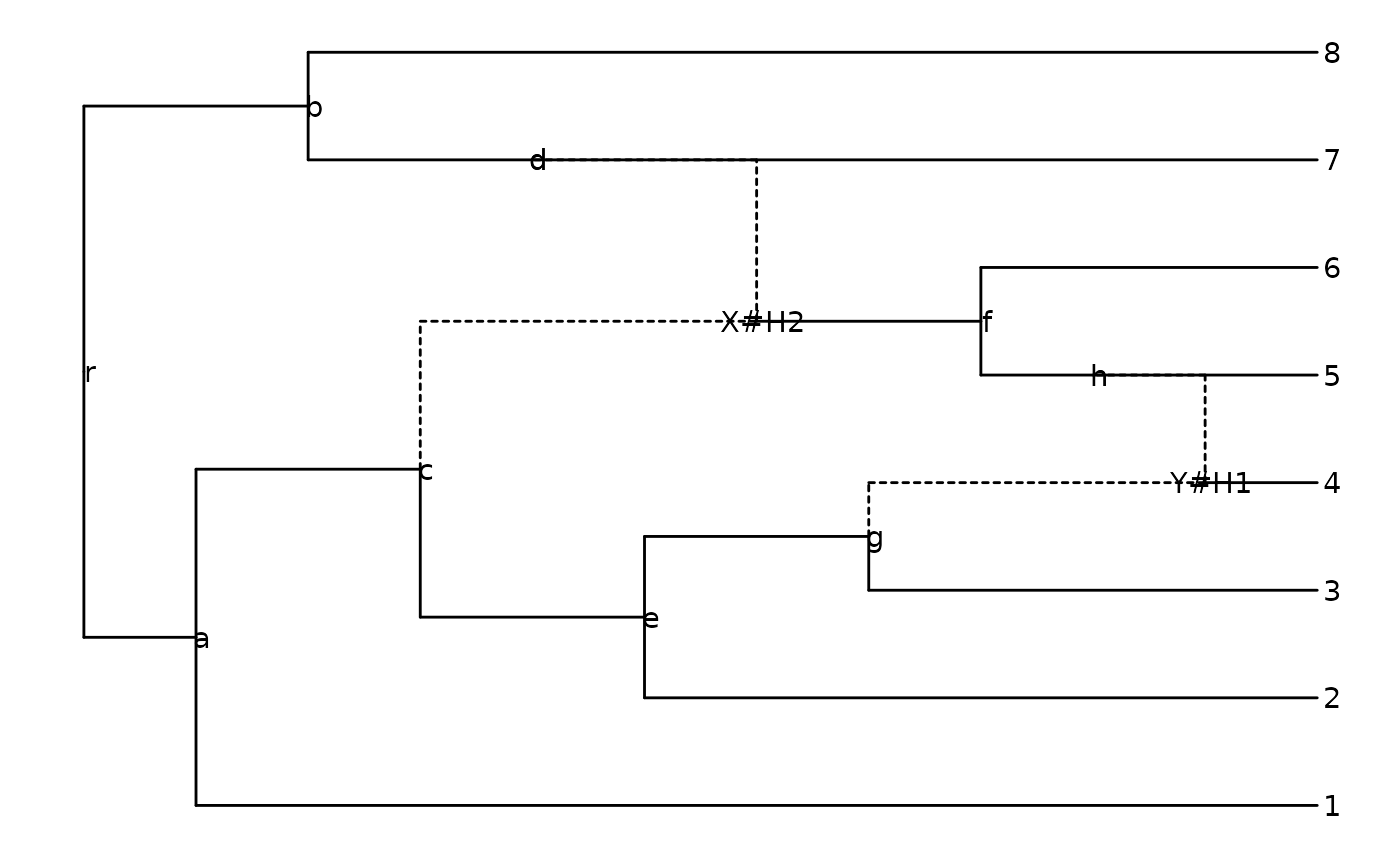
p <- ggevonet(z, layout = "slanted") + geom_tiplab() + geom_nodelab()
p + geom_tiplab(size=3, color="purple")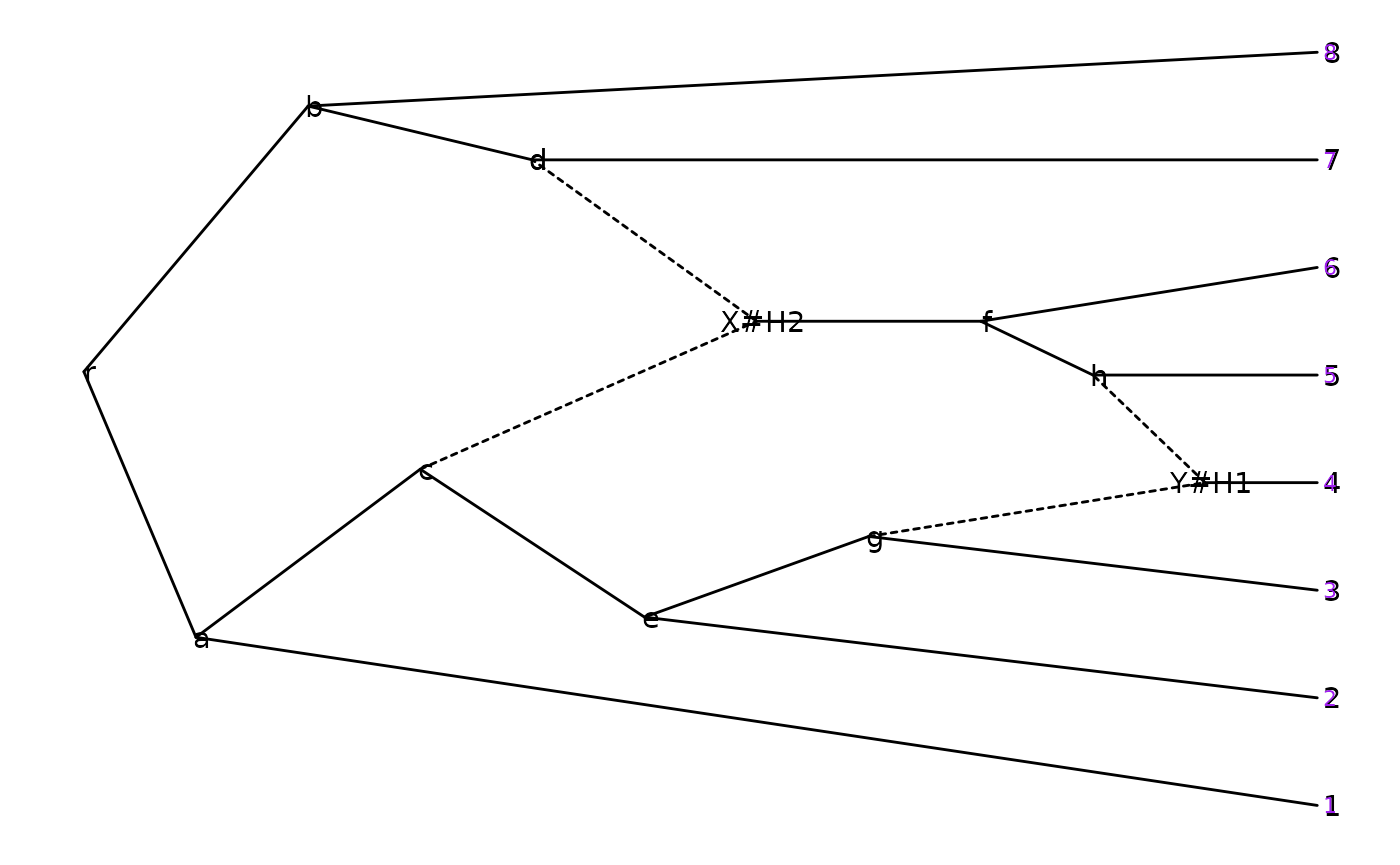
p + geom_nodepoint(color="#b5e521", alpha=1/4, size=10)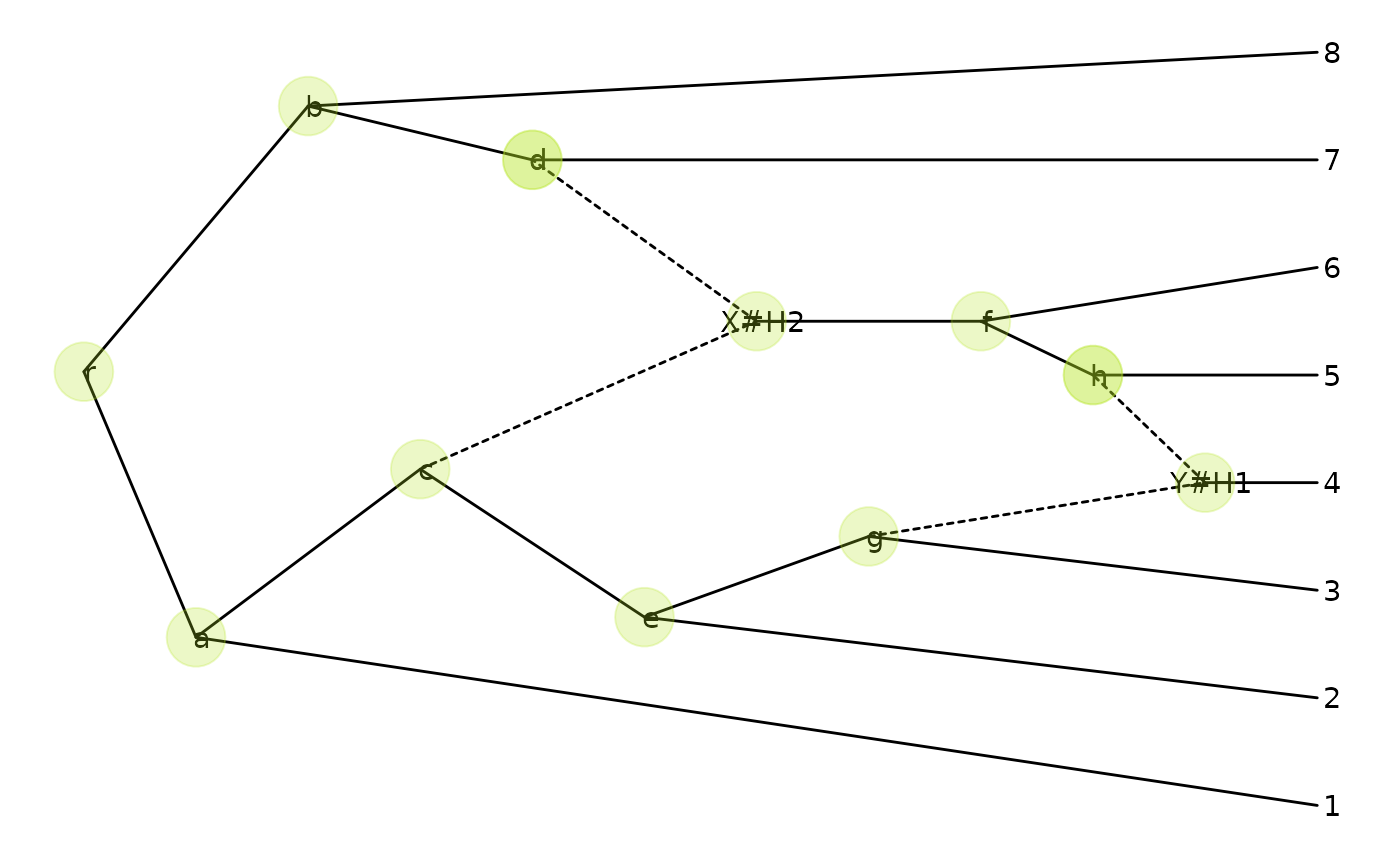
Resumen
Esta viñeta ilustra todas las funciones en el paquete tanggle para R. Aquí se proveen algunos ejemplos de como graficar redes implícitas y explícitas. La visualización de redes divididas toma (se sirve de / utiliza ???) la mayoría de las funciones compatibles con árboles no enraizados en ggtree. Las opciones de diseño para las redes explícitas son rectangular o slanted.
Session info
sessionInfo()
#> R version 4.5.1 (2025-06-13)
#> Platform: x86_64-pc-linux-gnu
#> Running under: Ubuntu 24.04.3 LTS
#>
#> Matrix products: default
#> BLAS: /usr/lib/x86_64-linux-gnu/openblas-pthread/libblas.so.3
#> LAPACK: /usr/lib/x86_64-linux-gnu/openblas-pthread/libopenblasp-r0.3.26.so; LAPACK version 3.12.0
#>
#> locale:
#> [1] LC_CTYPE=C.UTF-8 LC_NUMERIC=C LC_TIME=C.UTF-8
#> [4] LC_COLLATE=C.UTF-8 LC_MONETARY=C.UTF-8 LC_MESSAGES=C.UTF-8
#> [7] LC_PAPER=C.UTF-8 LC_NAME=C LC_ADDRESS=C
#> [10] LC_TELEPHONE=C LC_MEASUREMENT=C.UTF-8 LC_IDENTIFICATION=C
#>
#> time zone: UTC
#> tzcode source: system (glibc)
#>
#> attached base packages:
#> [1] stats graphics grDevices utils datasets methods base
#>
#> other attached packages:
#> [1] phangorn_2.12.1 ape_5.8-1 tanggle_1.15.3 ggtree_3.16.3
#> [5] ggplot2_4.0.0 BiocStyle_2.36.0
#>
#> loaded via a namespace (and not attached):
#> [1] yulab.utils_0.2.1 rappdirs_0.3.3 sass_0.4.10
#> [4] generics_0.1.4 tidyr_1.3.1 ggplotify_0.1.3
#> [7] lattice_0.22-7 digest_0.6.37 magrittr_2.0.4
#> [10] evaluate_1.0.5 grid_4.5.1 RColorBrewer_1.1-3
#> [13] bookdown_0.45 fastmap_1.2.0 Matrix_1.7-3
#> [16] jsonlite_2.0.0 BiocManager_1.30.26 purrr_1.1.0
#> [19] aplot_0.2.9 scales_1.4.0 codetools_0.2-20
#> [22] lazyeval_0.2.2 textshaping_1.0.4 jquerylib_0.1.4
#> [25] cli_3.6.5 rlang_1.1.6 tidytree_0.4.6
#> [28] withr_3.0.2 cachem_1.1.0 yaml_2.3.10
#> [31] tools_4.5.1 parallel_4.5.1 dplyr_1.1.4
#> [34] fastmatch_1.1-6 gridGraphics_0.5-1 vctrs_0.6.5
#> [37] R6_2.6.1 lifecycle_1.0.4 ggfun_0.2.0
#> [40] fs_1.6.6 htmlwidgets_1.6.4 treeio_1.32.0
#> [43] ragg_1.5.0 pkgconfig_2.0.3 desc_1.4.3
#> [46] pkgdown_2.1.3 pillar_1.11.1 bslib_0.9.0
#> [49] gtable_0.3.6 glue_1.8.0 Rcpp_1.1.0
#> [52] systemfonts_1.3.1 xfun_0.53 tibble_3.3.0
#> [55] tidyselect_1.2.1 knitr_1.50 farver_2.1.2
#> [58] igraph_2.2.0 patchwork_1.3.2 htmltools_0.5.8.1
#> [61] nlme_3.1-168 labeling_0.4.3 rmarkdown_2.30
#> [64] compiler_4.5.1 quadprog_1.5-8 S7_0.2.0